热图分析模型(热图怎么画)
导语:生信分析,什么是热图以及如何使用R画热图
什么是热图
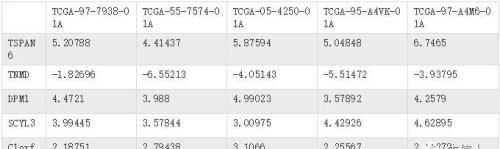
当我有一个二维矩阵,例如:
这是一个TCGA肺腺癌的基因表达谱(logCPM,前5个基因、5个样本)。我想以直观的方式观察这个二维矩阵,怎么办?
这个时候就要用到热图了。这个矩阵的热图(前50个基因、50个样本):
图一
细心的看官会看出来这个热图跟在文献中见到的热图有点不一样,在文献中见到的热图是这样的:
图二
这是为什么呢?原来在 图一 中直接看很难看出什么信息,有人就想啊,能不能对行和列进行排序,把相似的排到一起 这样不就能直观地反应信息了吗?当然排序的方法有很多,你可以自己排,也可以使用聚类的方法。所谓聚类的方法 就是对行列(可以都聚类,也可以只对行聚类,或者只对列聚类)进行聚类,把聚到同一类的放到一起。图二 就是对 样本和基因都进行了层次聚类,距离较近的放到一起。
R语言如何画热图
R语言能画热图的方法有很多,本文介绍如何使用 pheatmap 画热图。pheatmap 包很简单,整个包只有一个函数 pheatmap ,下面我们就来看看这个函数的用法。
导入包并加载数据
library(pheatmap)exp <- read.delim(, row.names = 1, check.names = FALSE)如何画出带聚类的热图
pheatmap(exp)如何画出不带聚类的热图
pheatmap(exp, cluster_rows = FALSE, cluster_cols = FALSE)br如果样本已经有分组结果该怎么展示
很多情况下,我们的样本已经分好组了。例如,现在我们的样本前25个属于 clsuter1 ,后25个属于cluster2 。
cls <- c(rep(, 25), rep(, 25))print(cls)br[1] [9] [17] [25] [33] [41] [49] br这个时候就要用到 annotation 参数了( annotation_row 或 annotation_col )。
使用 annotation 需要注意的是:
1.参数的值必须是 data.frame
2.data.frame 的行名必须跟需要 annotation 的行或列一致。
names(cls) <- colnames(exp)pheatmap(exp, cluster_cols = FALSE, annotation_col = data.frame(cls = cls))图三
annotation可不可以有多个
细心的看官又发现了,既然 annotation 是一个 data.frame ,那如果我的 data.frame 有多列会是什么效果呢?
比如,我不但有样本分组,我还知道每个样本的肺腺癌亚型:
subtype = c(rep(, 10), rep(, 40))names(subtype) = colnames(exp)pheatmap(exp, cluster_cols = FALSE, annotation_col = data.frame(cls = cls, subtype = subtype))br免责声明:本站部份内容由优秀作者和原创用户编辑投稿,本站仅提供存储服务,不拥有所有权,不承担法律责任。若涉嫌侵权/违法的,请反馈,一经查实立刻删除内容。本文内容由快快网络小珊创作整理编辑!